НАДН-дегидрогена́зный ко́мплекс, также называемый ко́мплекс I или НАДН-убихино́н-оксидоредукта́за — первый мультибелковый комплекс?! дыхательной цепи переноса электронов. Множество копий комплекса расположены в мембранах прокариотических организмов, способных к кислородному дыханию и внутренних мембранах митохондрий?! эукариотических клеток. По отношению к белкам человека комплекс I часто называют НАДН-дегидрогеназой.
| НАДН-убихинон-оксидоредуктаза | |
|---|---|
 Серым цветом показана внутренняя мембрана митохондрии. Сверху — митохондриальный матрикс, снизу — межмембранное пространство. |
|
| Идентификаторы | |
| Шифр КФ | 1.6.5.3 |
| Базы ферментов | |
| IntEnz | IntEnz view |
| BRENDA | BRENDA entry |
| ExPASy | NiceZyme view |
| MetaCyc | metabolic pathway |
| KEGG | KEGG entry |
| PRIAM | profile |
| PDB structures | RCSB PDB PDBe PDBj PDBsum |
| Gene Ontology | AmiGO • EGO |
| Поиск | |
| PMC | статьи |
| PubMed | статьи |
| NCBI | NCBI proteins |
Этот комплекс играет центральную роль в процессах клеточного дыхания и окислительного фосфорилирования: почти 40 % протонного градиента[en]* для синтеза АТФ создаются именно этим комплексом[1]. Комплекс I окисляет НАДН и восстанавливает одну молекулу убихинона, которая высвобождается в мембрану. На каждую окисленную молекулу НАДН комплекс переносит через мембрану четыре протона.
Комплекс І (НАДН-дегидрогеназа) был выделен из ряда объектов: митохондрий сердца быка, сахарной свеклы (Beta vulgaris), картофеля (Solanum tuberosum), бобов (Vicia faba), арабидопсиса (Arabidopsis thaliana) и риса (Oryza sativa), а также из митохондрий гриба нейроспоры Neurospora crassa и мембран кишечной палочки (Esherichia сoli)[2].
Содержание
- 1 Структурная организация комплекса I
- 2 Реакция
- 3 Эволюционное происхождение
- 4 Образование активных форм кислорода
- 5 Ингибиторы
- 6 Патологии
- 7 См. также
- 8 Примечания
- 9 Источники
- 10 Литература
- 11 Внешние ссылки
Структурная организация комплекса I

У прокариот комплекс І состоит из 14 основных субъединиц, образующих ядро комплекса, без которого он не функционирует. Семь субъединиц чрезвычайно гидрофобны и локализованы в мембране, а семь относительно гидрофильных расположены вне мембраны. У эукариот в результате эволюции комплекс покрылся «шубой» из приблизительно 30 вспомогательных субъединиц, их количество может варьировать в зависимости от объекта. Так, у млекопитающих этот фермент состоит из 44 субъединиц, а у гриба Yarrowia lipolytica — из 48[3]. В результате этой надстройки молекулярная масса комплекса I увеличилась почти вдвое: с ~550 кДа у бактерий до ~1 МДа в митохондриях[4].
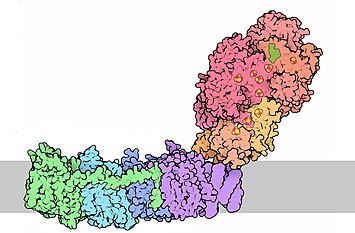
Электронная микроскопия показала, что комплекс I (как из бактерий, так и из митохондрий) имеет характерную L-образную форму. Из-за этой формы, а также необычной, как бы мятой молекулярной поверхности, комплекс I получил от учёных прозвище «старый башмак». Гидрофобная «подошва» представлена белками, встроенными в мембрану, а гидрофильная часть — «лодыжка» — обращена в матрикс?![2].
Четыре субъединицы убихинон-связывающего модуля совместно с субъединицами мембранной части фермента образуют сайт связывания убихинона, в котором он взаимодействует с железосерным кластером N2, принимает два электрона и восстанавливается до убихинола?!. Кластер N2, последний в ряду кластеров, по которым электроны от НАДН передаются к убихинону, приподнят над мембраной на ~15 Å. Сама полость, в которой связывается убихинон, имеет длину 30 Å и может вместить всю молекулу вместе с длинным гидрофобным хвостом из семи изопреновых единиц. Полость имеет узкий вход, так что длинная гидрофобная цепь вынуждена принять определённую конформацию, которая сохраняется на протяжении всей ферментативной реакции. Наличие такого длинного и узкого сайта связывания является характерной чертой комплекса I. Внутри полости убихинон взаимодействует с консервативными остатками тирозина и гистидина[5].
У грибов, животных и сосудистых растений по крайней мере семь из 44 субъединиц, которые образуют мембранный домен, кодируются митохондриальным геномом[6]. У млекопитающих этих субъединиц ровно семь[7][8]. У растений ДНК митохондрий кодирует девять субъединиц: кроме семи субъединиц, которые входят в состав гидрофобной части комплекса, в ней закодированы две субъединицы, гомологичные субъединицам 49 кДа и 30 кДа млекопитающих, а остальные компоненты находятся под контролем ядерных генов[2]. Однако эти данные, полученные при исследовании пластомов картофеля и арабидопсиса, могут оказаться неверны для других видов растений, а количество закодированных в митохондриях субъединиц может изменяться от вида к виду. Так, у печёночника Marchantia polymorpha субъединица NAD7, гомологичная полипептиду 49 кДа, кодируется ядерным геномом и транспортируется в митохондрии, а соответствующий ген митохондриальной ДНК превратился в псевдоген и не функционален[9].
Исследования показали, что по своим свойствам комплекс I существенно не отличается у объектов животного и растительного происхождения[2]. Однако у растений имеются некоторые специфические субъединицы, которые в некоторых случаях приводят к функциональным особенностям. Анализ комплекса I у Arabidopsis показывает, что более 30 % субъединиц специфичны только для растений[10]. Например, частью мембранного модуля растительного комплекса I является так называемый γ-карбоангидразный структурный модуль и L-галактоно-1,4-лактондегидрогеназа, которая в то же время представляет собой последний фермент митохондриального пути биосинтеза аскорбиновой кислоты[11].
Таблица основных (коровых) субъединиц
| Коровые субъединицы[4][12][5] | ||||||
|---|---|---|---|---|---|---|
| Корова | Человек | Кишечная палочка | Арабидопсис | Описание | Кофакторы | |
| Периферическая рука | ||||||
| Дегидрогеназный модуль | 75 кДа | NDUFS1 | NuoG | 75 кДа | транспорт электронов | Fe2S2 (N1b), 2 x Fe4S4 (N4 и N5), (N7)[a 1] |
| 51 kDa | NDUFV1 | NuoF | 51 кДа | связывание НАДН, транспорт электронов | флавин, Fe4S4 (N3) | |
| 24 kDa | NDUFV2 | NuoE | 24 кДа | Несёт вспомогательный кластер N1a | Fe2S2 (N1a) | |
| Убихинон-связывающий модуль | 49 кДа | NDUFS2 | NuoD (NuoCD)[a 2] | NAD7 | ||
| 30 kDa | NDUFS3 | NuoC | NAD9 | |||
| TYKY | NDUFS8 | NuoI | 28,5 кДа | транспорт электронов | 2 x Fe4S4 (N6a и N6b) | |
| PSST | NDUFS7 | NuoB | 20 кДа | транспорт электронов | Fe4S4 (N2) | |
| Мембранная рука | ||||||
| — | ND1 | MT-ND1 | NuoH | NAD1 | ||
| Протон-транслоцирующий модуль | ND2 | MT-ND2 | NuoN | NAD2 | антипортер-подобный белок | |
| ND3 | MT-ND3 | NuoA | NAD3 | |||
| ND4 | MT-ND4 | NuoM | NAD4 | антипортер-подобный белок | ||
| ND4L | MT-ND4L | NuoK | NAD4L | |||
| ND5 | MT-ND5 | NuoL | NAD5 | антипортер-подобный белок | ||
| ND6 | MT-ND6 | NuoJ | NAD6 | |||
Таблица вспомогательных субъединиц
Все митохондриальные комплексы I имеют множество дополнительных субъединиц, которые не являются необходимыми для каталитической активности и отличаются у разных видов. Очевидно, что они несут определённую функциональную нагрузку, поскольку мутации в них приводят к врождённым заболеваниям. Для некоторых субъединиц показано наличие определённых функций, так B16.6 (GRIM-19) участвует в апоптозе, а субъединица 39 кДа (NDUFA9) — в регуляции активности комплекса[13]. Что касается остальных субъединиц, то сейчас активно обсуждается их возможная роль в регуляции, сборке, стабилизации и защите от активных форм кислорода. Следует упомянуть, что дополнительные субъединицы значительно увеличивают энергетические затраты клетки на синтез, сборку и деградацию комплекса. Такие затраты, тем не менее, могут окупится в случае эукариотической клетки, в которой процесс синтеза белка хорошо контролируется и в этом смысле доведён до совершенства. С другой стороны, если вспомогательные субъединицы необходимы для стабилизации комплекса I, остаётся непонятным каким образом бактериальные комплексы, которые состоят из минимально необходимого количества полипептидов, успешно функционируют без них. На данный момент у учёных нет чёткого ответа на эти вопросы[5].
| Вспомогательные субъединицы[4][12][5][14] | ||||
|---|---|---|---|---|
| No. | Корова | Белок человека | Описание | Семейство белков Pfam |
| Субъединицы | ||||
| 15 | 13A | NDUFS6 | участвует в сборке и стабилизирует комплекс | Pfam PF10276 |
| 16 | B17.2 | NDUFA12 | Pfam PF05071 | |
| 17 | AQDQ | NDUFS4 | возможно фосфорилируется ПКА, стабилизирует комплекс во время сборки |
Pfam PF04800 |
| 18 | 39kDa | NDUFA9 | соединяет мембранный и периферический домены, участвует в активации/деакцтивации комплекса |
Pfam PF01370 |
| 19 | ACPM | ACPM_HUMAN | ацил-переносящий белок | Pfam PF00550 |
| 20 | B8 | NDUA2_HUMAN | Pfam PF05047 | |
| 21 | MFWE | NDUFA1 | цАМФ зависимое фосфорилирование ПКА | Pfam PF15879 |
| 22 | B12 | NDUFB3 | Pfam PF08122 | |
| 23 | AB13 | NDUFA5 | участвует в активации/деакцтивации комплекса | Pfam PF04716 |
| 24 | B14 | NDUFA6 | Pfam PF05347 | |
| 25 | B14.7 | NDUFA11 | Pfam PF02466 | |
| 26 | ESSS | NDUFB11 | цАМФ зависимое фосфорилирование ПКА | Pfam PF10183 |
| 27 | PFFD | NDUFS5 | Pfam PF10200 | |
| 28 | B15 | NDUFB4 | Pfam PF07225 | |
| 29 | B16.6 | NDUFA13 | участвует в апоптозе | Pfam PF06212 |
| 30 | B18 | NDUFB7 | Pfam PF05676 | |
| 31 | PGIV | NDUFA8 | Pfam PF06747 | |
| 32 | B22 | NDUFB9 | Pfam PF05347 | |
| 33 | PDSW | NDUFB10 | фосфорилируется Src в раковых клетках | Pfam PF10249 |
| 34 | ASHI | NDUFB8 | Pfam PF05821 | |
| 35 | B14.5B | NDUFC2 | в норме фосфорилирована | Pfam PF06374 |
| 36 | AGGG | NDUFB2 | Pfam PF14813 | |
| 37 | B14.5A | NDUFA7 | в норме фосфорилирована | Pfam PF07347 |
| 38 | B9 | NDUFA3 | Pfam PF14987 | |
| 39 | MLRQ[a 3] | NDUA4_HUMAN | Pfam PF06522 | |
| 40 | SGDH | NDUFB5 | Pfam PF09781 | |
| 41 | MNLL | NDUFB1 | Pfam PF08040 | |
| 42 | KFYI | NDUFC1 | в норме фосфорилирована | Pfam PF15088 |
| 43 | 42kD | NDUFA10 | Pfam PF01712 | |
| 44 | NDUFA4L2 | NDUFA4L2 | Pfam PF15880 | |
| 45 | NDUFV3 | NDUFV3 | — | |
| 46 | NDUFB6 | NDUFB6 | Pfam PF09782 | |
| Вспомогательные белки, осуществляющие сборку[15] |
||||
| 47 | NDUFAF1[a 3] | CIA30_HUMAN | фактор сборки комплекса 1 | Pfam PF08547 |
| 48 | NDUFAF2 | MIMIT_HUMAN | фактор сборки комплекса 2 | Pfam PF05071 |
| 49 | NDUFAF3 | NDUF3_HUMAN | фактор сборки комплекса 3 | Pfam PF05071 |
| 50 | NDUFAF4 | NDUF4_HUMAN | фактор сборки комплекса 4 | Pfam PF06784 |
Некоторые дополнительные субъединицы фосфорилируются различными киназами, чего никогда не происходит с коровыми субъединицами. Предположителагается, что таким образом происходит регуляция работы комплекса. Как одна из субъединиц в комплексе присутствует ацил-переносящий белок (NDUFAB1) с фосфорилированной пантотеновой кислотой в качестве в простетической группы. Предполагают, что он участвует в синтезе липоевой кислоты, репарации повреждённых липидов мембраны или модифицирует другие белки остатками миристиновой кислоты. Следует отметить, что функционирование этого белка не зависит от прямого физического контакта с комплексом I и значительная его часть присутствует в свободном виде внутри митохондриального матрикса[14].
Кофакторы
 Электрон-транспортная цепь комплекса I. Серые стрелочки — маловероятный или ныне не существующий путь переноса
Электрон-транспортная цепь комплекса I. Серые стрелочки — маловероятный или ныне не существующий путь переноса
Все простетические группы НАДН-дегидрогеназного комплекса (один флавинмононуклеотид и от 8 до 9 железосерных кластеров) находятся в периферическом водорастворимом домене. У млекопитающих, как и у всех позвоночных, их восемь[7]. Семь кластеров образуют электрон-транспортную цепь длинной в ~96 Å от ФМН до места связывания убихинона. На основе современных данных считается, что перенос электрона происходит по следующему пути: НАДН → ФМН → N3 → N1b → N4 → N5 → N6a → N6b → N2 → Q. Сначала происходит передача двух электронов на флавин, а затем они по одному передаются через цепочку кластеров к сайту связывания хинона и восстанавливают его до состояния Q−2. Кластер N1a располагается недалеко от флавинового кофактора и на некотором расстоянии от магистральной цепи переноса электрона. Этот кластер высококонсервативен у разных видов; полагают, что он осуществляет контроль скорости транспорта электрона внутри комплекса, перебрасываясь электроном с ФМН[4]. Существует модель, согласно которой один из электронов с флавина идёт по магистральному пути на хинон, а другой запасается в кластере N1a и позже возвращается в основную цепь, через флавосемихинон. Возможно, такой механизм позволяет снизить образование активных форм кислорода на восстановленном флавине. К тому же, это позволяет стабилизировать (до миллисекунды) состояние, когда последний кластер N2 восстановлен, но нет второго электрона, чтобы завершить восстановление убихинона. Такое состояние может быть необходимым для конформационных изменений, сопряжённых с транспортом протонов.
Часть кластеров в цепи (N3, N4 и N6a) имеют высокий окислительно-восстановительный потенциал (редокс-потенциал) на уровне —0,25 В, в то время как три других (N1b, N5 и N6b) имеют более низкие потенциалы. В результате редокс-потенциал на пути электрона меняется наподобие американских горок. Такая кривая изменения энергетического состояния характерна для многих окислительно-восстановительных ферментов: она позволяет оптимизировать скорость транспорта электрона и добиться эффективного переноса энергии[4].
Кластер N5 имеет очень низкий потенциал и лимитирует скорость общего потока электронов по всей цепи. Вместо обычных для железосерных центров лигандов (четырёх остатков цистеина) он скоординирован тремя остатками цистеина и одним остатком гистидина, а также окружён заряженными полярными остатками, хотя и находится в глубине фермента[4].
Необычные лиганды имеет и терминальный кластер цепи — N2. Его редокс-потенциал самый высокий из всех кластеров (от —0,1 до —0,15 В). Он связан с четырьмя последовательно расположенными в полипептидной цепи остатками цистеина, что создаёт напряжённую конформацию. Из-за этого при его восстановлении происходят конформационные изменения соседних цепей, возможно, связанные с транспортом протона[4].
Кластер N7 присутствует только в комплексе I некоторых бактерий. Он значительно удалён от остальных кластеров и не может обмениваться с ними электронами, так что по-видимому, является реликтом. В некоторых бактериальных комплексах, родственных комплексу I, между N7 и остальными кластерами обнаружены четыре консервативных остатка цистеина, а комплексе I бактерии Aquifex aeolicus был обнаружен дополнительный Fe4S4 кластер, соединяющий N7 с остальными кластерами. Из этого следует вывод, что у A. aeolicus комплекс I, кроме НАДН, может использовать иной донор электронов, который передаёт их через N7[18].
Сборка митохондриального комплекса I
Митохондриальный комплекс I образует с дыхательными комплексами III и IV суперкомплексы, именуемые респирасомами. В митохондриях млекопитающих и человека около 90 % комплекса находится в составе респирасом. Также на митохондриях из молодых корневищ бамбука показано, что 90 % от общего количества комплекса I собрано в респирасомы, а у Arabidopsis — в суперкомплекс I—III2[19]. Накоплено достаточно доказательств того, что присутствие респирасом необходимо для стабильности и функционирования комплекса I, который в отсутствии комплексов III или IV нестабилен. Например, на мутантных клетках человека показано, что комплекс I необходим для формирования комплекса III, и с другой стороны, утрата комплекса III приводит к потере комплекса I. Кроме того, в ряде исследований на животных клетках приводятся доказательства того, что для стабильности комплекса I необходимы комплексы IV и димер комплекса III.
Недавно на культуре клеток человека было показано, что комплексы IV и III необходимы для сборки полноценного комплекса I, при этом сам не до конца собранный комплекс служит основой для образования респирасом. Наличие в респирасоме комплексов IV и III нужно для присоединения к комплексу I каталитических субъединиц НАДН-дегидрогеназного модуля, которые полностью активируют комплекс и всю респирасому[20].
Реакция
НАДН-дегидрогеназный комплекс окисляет НАДН, образовавшийся в матриксе в ходе цикла трикарбоновых кислот. Электроны от НАДН используются для восстановления мембранного переносчика, убихинона Q, который переносит их к следующему комплексу электрон-транспортной цепи митохондрий, комплексу III или цитохром-bc1-комплексу[21].
НАДН-дегидрогеназный комплекс работает как протонная помпа: на каждый окисленный НАДН и восстановленный Q через мембрану в межмембранное пространство перекачиваются четыре протона[22]:
- НАДН + H+ + Q + 4H+in → НАД+ + QH2 + 4H+out
Образовавшийся в ходе реакции электрохимический потенциал используется для синтеза АТФ. Любопытно, что реакция, катализируемая комплексом I, обратима, этот процесс называется аэробное сукцинат-индуцированное восстановление НАД+. В условиях большого потенциала на мембране и избытка восстановленных убихинолов комплекс может восстанавливать НАД+ с использованием их электронов и пропускать протоны обратно в матрикс. Этот феномен обычно наблюдается, когда много сукцината, но мало оксалоацетата или малата. Восстановление убихинона осуществляется ферментами сукцинатдегидрогеназой, глицерол-3-фосфатдегидрогеназой[en] или митохондриальной дигидрооротатдегидрогеназой[en]. В условиях высокого протонного градиента сродство комплекса к убихинолу повышается, а редокс-потенциал убихинола снижается благодаря росту его концентрации, что и делает возможным обратный транспорт электронов по электрическому потенциалу внутренней мембраны митохондрий к НАД[23]. Данный феномен удалось наблюдать в лабораторных условиях, но неизвестно, имеет ли он место в живой клетке.
Механизм транспорта протонов
На начальных этапах исследования комплекса I широко обсуждалась модель, основанная на предположении, что в комплексе оперирует система, похожая на Q-цикл[en]. Однако позднейшие исследования не обнаружили в комплексе I каких-либо внутренне связанных хинонов и полностью опровергли эту гипотезу[24].
НАДН-дегидрогеназный комплекс, по-видимому, имеет уникальный механизм транспорта протонов посредством конформационных изменений самого фермента. Субъединицы ND2, ND4 и ND5 называются антипорт-подобными, поскольку они гомологичны друг другу и бактериальным Mrp Na+/H+ антипортам. Эти три субъединицы образуют три основных протонных канала, которые состоят из консервативных остатков заряженных аминокислот (в основном лизина и глутамата). Четвёртый протонный канал образован частью субъединицы Nqo8 и малыми субъединицами ND6, ND4L и ND3. Канал сходен по строению с аналогичными каналами антипорт-подобных субъединиц, но содержит необычно много плотно упакованных остатков глутамата со стороны матрикса, за что и получил название E-канал (латинское E используется как стандартное обозначение глутамата). От С-конца субъединицы ND5 отходит удлинение, состоящее из двух трансмембранных[en] α-спиралей, соединённых необычно протяжённой (110 Å) α-спиралью[4] (HL), которая, проходя по стороне комплекса, обращённой в матрикс, физически соединяет все три антипорт-подобные субъединицы, и возможно, участвует в сопряжении транспорта электронов с конформационной перестройкой. Ещё один сопрягающий элемент, βH, образован серией перекрывающихся β-шпилек?! и α-спиралей, он расположен на противоположной, периплазматической стороне комплекса[25].
До сих пор окончательно неизвестно, как именно транспорт электронов сопряжён с переносом протонов. Полагают, что мощный отрицательный заряд кластера N2 может расталкивать окружающие полипептиды, вызывая тем конформационные изменения, которые неким образом распространяются на все антипорт-подобные субъединицы, расположенные довольно далеко друг от друга. Другая гипотеза предполагает, что изменение конформации вызывает в необычно длинном сайте связывания убихинона стабилизированный убихинол Q−2 с крайне низким редокс-потенциалом и отрицательным зарядом. Неизвестными остаются и многие детали кинетики конформационных изменений и сопряжённого с ними транспорта протонов[25].
Активная и неактивная формы
 |
 |
|||||||||
| Комплекс I из быка и Yarrowia lipolytica | ||||||||||
Эукариотический НАДН-дегидрогеназный комплекс существует в двух чётко различимых формах: одна полностью функционирующая, так называемая активная или A-форма, и вторая, каталитически неактивная или D-форма. Если фермент находится при повышенных, но всё ещё физиологических температурах (> 30 °C) в отсутствие субстрата, фермент переходит в D-форму. Она каталитически неактивна, но может активироваться субстратом (НАДН и убихиноном, на который можно сбрасывать электроны). После одного или нескольких ферментативных циклов комплекс становится активным, а скорость реакции возрастает. Такой переход обнаружен только у позвоночных и грибов, но не у беспозвоночных или бактерий. Растительные комплексы не исследовались. В присутствии бивалентных катионов (Mg2+, Ca2+) или в щелочном pH активация происходит значительно дольше, а свободная пальмитиновая кислота сильно увеличивает частоту перехода из активной в деактивированную форму[26].
Высокая энергия активации (270 кДж/моль) перехода из A- в D-форму указывает, что в комплексе происходит значительная конформационная перестройка. До настоящего момента единственным выявленным различием между двумя формами остаётся количество остатков цистеина на поверхности фермента. Согласно последним данным, в этом процессе участвуют субъединицы, расположенные рядом с хинон-связывающим сайтом: 39 кДа, ND3 и ND1[26]. Обработка D-форм комплекса I специальными реагентами (N-этилмалеимидом или реактивом Эллмана) необратимо блокирует эти важные остатки цистеина, делая невозможным повторную активацию фермента. Любопытно, что A-форма комплекса I нечувствительна к сульфгидрилам, что указывает на то, что остатки цистеина спрятаны глубоко в белке. В свою очередь, деактивированная форма восприимчива к ингибированию нитрозотиолами[en] и пероксинитритом[27].
Конформационные изменения комплекса I имеют большое физиологическое значение. После гипоксии восстановление уровня кислорода может привести к всплеску окисления НАД(Ф)Н и генерации активных форм кислорода, которые могут повредить митохондрии и вызвать некроз тканей. Переход из активной в неактивную форму комплекса происходит при патологических состояниях, когда число оборотов фермента снижено при нормальной, физиологической температуре тела, например при гипоксии, ишемии или повышении соотношения оксид азота (NO)/кислород в тканях (так называемая метаболическая гипоксия). Таким образом, комплекс I предотвращает окисление остальных дыхательных комплексов, когда происходит восстановление уровня кислорода. Кроме того, неактивная форма не способна к обратному транспорту электронов, что снижает образование АФК[28][26].
Эволюционное происхождение
 Комплекс I из T. thermophilus
Комплекс I из T. thermophilus Кофакторы комплекса I
Кофакторы комплекса I
НАДН-дегидрогеназный комплекс относится к семейству мембранных оксидоредуктаз из класса NiFe-гидрогеназ[en], которые у анаэробных бактерий и архей сопрягают реакцию окисления субстрата и восстановления водорода с транспортом протонов. На основе данных о гомологии белков можно сделать вывод, что комплекс возник в результате объединения двух ранее существовавших комплексов из разных, неродственных белковых семейств. НАДН-дегидрогеназный и убихинон-связывающий модули произошли из растворимой NiFe-гидрогеназы, которая окисляла НАДН и восстанавливала водород, а гидрофобная мембранная «подошва» комплекса, перекачивающая протоны, возникла из Na+/H+-антипортов Mrp[4].
Слияние растворимой гидрогеназы и белков-антипортов привело к возникновению большого количества мембранных гидрогеназ и дегидрогеназ, которые могли позднее эволюционировать в комплекс I. Трёхмерная структура этих ферментов, вероятно, схожа с таковой у комплекса I. К дегидрогеназам можно отнести архейный комплекс Fpo из 11 субъединиц, который окисляет кофактор F420, связанный с водородом, и восстанавливает метанофеназин (аналог убихинона), перекачивая через мембрану один протон на два электрона. У этого фермента нет НАДН-дегидрогеназного модуля. К группе гидрогеназ относятся формиатводородлиазы из Escherichia coli: формиатводородлиаза-1 из семи субъединиц и формиатводородлиаза-2 из десяти. Оба фермента окисляют формиат, восстанавливая водород с переносом нескольких протонов через мембрану[18].
Самый простой из белков-родственников комплекса I — гидрогеназа Ech (англ. E. coli hydrogenase-3-type hydrogenase) археи Methanosarcina barkeri. Она состоит из всего шести субъединиц и перекачивает один протон в результате окисления ферредоксина с восстановлением молекулы водорода. Ech содержит минимальный набор субъединиц (гомологичных комплексу I), необходимый для сопряжения реакции окисления с транспортом протонов[18].
Кроме того, комплекс I обнаружен в хлоропластах в виде НАДН-дегидрогеназного комплекса хлоропластов. Его точное стороение и функция пока неизвестны[29].
Образование активных форм кислорода
Комплекс I в процессе своей работы образует активные формы кислорода[30]. Обычно это супероксид (а также пероксид водорода), и образуется он по крайней мере двумя путями. В ходе прямого транспорта электронов, в процессе дыхания, образуется очень малое количество супероксида (вероятно, менее 0,1 % от всего потока электронов переносятся на кислород)[30][31].
Во время обратного транспорта электронов, который происходит в условиях аэробного сукцинат-индуцированного восстановление НАД+, комплекс I, возможно, становится самым активным местом образования супероксида: до 5 % электронов идут на восстановление кислорода[32].
Супероскид образуется в НАДН-дегидрогеназный комплексе в результате переноса одного электрона с ФМНН2 к O2. Образовавшийся в результате радикал флавина нестабилен и переносит оставшийся электрон на железосерные кластеры. Уровень образования супероксида определяется соотношением НАДН/НАД+; в условиях когда восстановлено небольшое количество НАД, НАД+ успешно конкурирует за электроны с кислородом[33][34].
Ингибиторы
Наиболее изученный ингибитор комплекса I — ротенон (широко применяемый как органический пестицид). Ротенон и ротеноиды — это изофлавоноиды, которые присутствуют в корнях нескольких родов тропических растениях таких как Антония (Loganiaceae), Derris и Lonchocarpus (Fabaceae). Ротенон давно используется как инсектицид и рыбный яд, так как митохондрии насекомых и рыб особенно к нему чувствительны. Известно, что коренные жители Французской Гвианы и другие индейцы Южной Америки использовали ротенон-содержащие растения для рыболовства уже в XVII веке[35]. Ротенон взаимодействует с сайтом связывания убихинона и конкурирует с основным субстратом. Было показано, что долгосрочное системное подавление комплекса I ротеноном может индуцировать селективное отмирание дофаминергических нейронов (секретирующих в качестве нейротрансмиттера дофамин)[36]. Схожим образом действует и пиерицидин А, ещё один мощный ингибитор комплекса I, структурно схожий с убихиноном. К этой же группе относится и амитал натрия — производное барбитуровой кислоты[2].
Несмотря на более чем 50-летнее изучение комплекса I, так и не удалось обнаружить ингибиторы, блокирующие перенос электронов внутри комплекса. Гидрофобные ингибиторы, такие как ротенон или пиерицидин, просто прерывают перенос электрона с терминального кластера N2 на убихинон[36].
Ещё одно вещество, блокирующее комплекс I — это аденозиндифосфатрибоза, конкурентный ингибитор[en] в реакции окисления НАДН. Он связывается с ферментом в сайте связывания нуклеотида (ФАД)[37].
К одним из самых сильных ингибиторов комплекса I относится семейство ацетогенинов. Показано, что эти вещества образуют химические сшивки с субъединицей ND2, что косвенно указывает на роль ND2 в связывании убихинона[38]. Любопытно отметить, что ацетогенин роллиниастатин-2 стал первым из обнаруженных ингибиторов комплекса I, который связывается в другом месте, нежели ротенон[39].
Умеренным ингибиторным эффектом обладает антидиабетический препарат метформин; по-видимому, данное свойство препарата лежит в основе механизма его действия[40].
Патологии
Мутации в генах субъединиц комплекса I могут привести к митохондриальным заболеваниям, например, к синдрому Лея. Точечные мутации митохондриальных субъединиц этого комплекса также могут стать причиной оптической нейропатии Лебера. Есть доказательства, что дефекты в структуре комплекса I могут играть роль в этиологии болезни Паркинсона, возможно, из-за образования активных форм кислорода[41]. Так, было показано, что у культур клеток больных болезнью Паркинсона увеличена утечка протонов в комплексе I, что снижает максимальную дыхательную ёмкость[42]. У растений мутации по комплексу І описаны у табака (Nicotiana silvestris) и кукурузы (Zea mays): мутации сопровождались патологией пыльцы и приводили к цитоплазматической мужской стерильности[2].
Недавние исследования выявили необычную роль комплекса I в работе мозга. Активность этого фермента значительно снижена у пациентов с биполярным расстройством, но остаётся в норме у пациентов с депрессией или шизофренией. У пациентов с биполярным расстройством в префронтальной коре наблюдалось повышенное окисление и нитрирование белков. Эти результаты делают комплекс I мишенью для будущих терапевтических исследований биполярного расстройства[43][44].
Воздействие пестицидов, блокирующих комплекс I, может приводить к далеко идущим последствиям. Например, продолжительное воздействие низких концентраций органофосфата и пестицида дихлофоса вызывает дисфункцию печени. Дихлофос изменяет активность комплексов I и II, что приводит к замедлению транспорта электронов и снижению синтеза АТФ[45].
См. также
Примечания
- ↑ Железосерный кластер N7 есть только у некоторых бактерий (например у E. coli и T. thermophilus).
- ↑ У E. coli и некоторых других бактерий субъединицы NuoC (30 кДа) и NuoD (49 кДа) слиты в одну.
- ↑ 1 2 Обнаружена у некоторых грибов, таких как Schizosaccharomyces pombe.
Источники
- ↑ Rouslan G. Efremov, Rozbeh Baradaran & Leonid A. Sazanov (27 May 2010). “The architecture of respiratory complex I”. nature. 465: 441—445. DOI:10.1038/nature09066. PMID 20505720.
- ↑ 1 2 3 4 5 6 Ермаков, 2005, с. 237.
- ↑ Carroll J, Fearnley IM, Skehel JM, Shannon RJ, Hirst J, Walker JE (October 2006). “Bovine complex I is a complex of 45 different subunits”. J. Biol. Chem. 281 (43): 32724—7. DOI:10.1074/jbc.M607135200. PMID 16950771.
- ↑ 1 2 3 4 5 6 7 8 9 Leonid A. Sazanov (2015 Jun). “A giant molecular proton pump: structure and mechanism of respiratory complex I”. Nature Reviews Molecular Cell Biology. 16 (6): 375–388. DOI:10.1038/nrm3997. PMID 25991374. Проверьте дату в |date= (справка на английском)
- ↑ 1 2 3 4 Judy Hirst (June 2013). “Mitochondrial Complex I”. Annual Review of Biochemistry. 82: 551–575. DOI:10.1146/annurev-biochem-070511-103700.
- ↑ Cardol P, Lapaille M, Minet P, Franck F, Matagne RF, Remacle C. (2006 Sep). “ND3 and ND4L subunits of mitochondrial complex I, both nucleus encoded in Chlamydomonas reinhardtii, are required for activity and assembly of the enzym”. Eukaryot Cell. 5 (9): 1460–7. PMID 16963630. Проверьте дату в |date= (справка на английском)
- ↑ 1 2 Voet, Judith G.; Voet, Donald. Biochemistry. — 3rd. — New York : J. Wiley & Sons, 2004. — P. 813–826. — ISBN 0-471-19350-X.
- ↑ Balsa E, Marco R, Perales-Clemente E, Szklarczyk R, Calvo E, Landázuri MO, Enríquez JA (September 2012). “NDUFA4 is a subunit of complex IV of the mammalian electron transport chain”. Cell Metab. 16 (3): 378—86. DOI:10.1016/j.cmet.2012.07.015. PMID 22902835.
- ↑ Allan G Rasmussonb, Volker Heiserc, Eduardo Zabaletaa, Axel Brennickea, Lutz Grohmannd (May 1998). “Physiological, biochemical and molecular aspects of mitochondrial complex I in plants”. Biochimica et Biophysica Acta (BBA) — Bioenergetics. 1364 (2): 101—111. DOI:10.1016/S0005-2728(98)00021-8.
- ↑ Peters K., Belt K., Braun H.-P. (2013). “3D gel map of Arabidopsis complex I”. Front. Plant Sci. Plant Proteomics. 5 (153). DOI:10.3389/fpls.2013.00153. PMID 23761796.
- ↑ Meyer E.H. (2012 May 24). “Proteomic investigations of complex I composition: how to define a subunit?”. Front. Plant Sci. Plant Proteomics. 3 (106). DOI:10.3389/fpls.2012.00106. PMID 22654890. Проверьте дату в |date= (справка на английском)
- ↑ 1 2 Cardol P (2011). “Mitochondrial NADH:ubiquinone oxidoreductase (complex I) in eukaryotes: a highly conserved subunit composition highlighted by mining of protein databases”. Biochim Biophys Acta. 1807 (11): 1390—7. DOI:10.1016/j.bbabio.2011.06.015. PMID 21749854.
- ↑ Marion Babot, Amanda Birch, Paola Labarbuta, Alexander Galkin (July 2014). “Characterisation of the active/de-active transition of mitochondrial complex I”. Biochimica et Biophysica Acta (BBA) — Bioenergetics. 1837 (7): 1083—1092. DOI:10.1016/j.bbabio.2014.02.018. PMID 24569053.
- ↑ 1 2 Katarzyna Kmita, Volker Zickermann (Oct 01, 2013,). “Accessory subunits of mitochondrial complex I”. Biochemical Society Transactions. 41 (5): 1272–1279. DOI:10.1042/BST20130091. Проверьте дату в |year= (справка на английском)
- ↑ Ogilvie I, Kennaway NG, Shoubridge EA (2005). “A molecular chaperone for complex I assembly is mutated in a progressive encephalopathy”. J Clin Invest. 115 (10): 2784—92. DOI:10.1172/JCI26020. PMC 1236688. PMID 16200211.
- ↑ Dunning CJ, McKenzie M, Sugiana C, Lazarou M, Silke J, Connelly A; et al. (2007). “Human CIA30 is involved in the early assembly of complex I and mutations in its gene cause disease”. EMBO J. 26 (13): 3227—37. DOI:10.1038/sj.emboj.7601748. PMC 1914096. PMID 17557076.
- ↑ Saada A, Vogel RO, Hoefs SJ, van den Brand MA, Wessels HJ, Willems PH; et al. (2009). “Mutations in NDUFAF3 (C3ORF60), encoding an NDUFAF4 (C6ORF66)-interacting complex I assembly protein, cause fatal neonatal disease”. Am J Hum Genet. 84 (6): 718—27. DOI:10.1016/j.ajhg.2009.04.020. PMC 2694978. PMID 19463981.
- ↑ 1 2 3 Rouslan G. Efremov, Leonid A. Sazanov (October 2012). “The coupling mechanism of respiratory complex I — A structural and evolutionary perspective”. Biochimica et Biophysica Acta. 1817 (10): 1785—1795. DOI:10.1016/j.bbabio.2012.02.015. PMID 22386882.
- ↑ Eubel H., Jänsch L., Braun H.P. (2003). “New insights into the respiratory chain of plant mitochondria: supercomplexes and a unique composition of complex II”. Plant Physiol. 133: 274—286. DOI:10.1104/pp.103.024620 Проверьте параметр |doi= (справка на английском). PMID 12970493. Символ zero width space в |doi= на позиции №4 (справка)
- ↑ David Moreno-Lastres, Flavia Fontanesi, Inés García-Consuegra, Miguel A. Martín, Joaquín Arenas, Antoni Barrientos, and Cristina Ugalde1 (Mar 7). “Mitochondrial Complex I plays an Essential Role in Human Respirasome Assembly”. Cell Metab. 15 (3): 324—335. DOI:10.1016/j.cmet.2012.01.015. Проверьте дату в |date= (справка на английском)
- ↑ Berg, J, Tymoczko, J, and L Stryer. Biochemistry. — 6th. — New York : WH Freeman & Company, 2006. — P. 509–513.
- ↑ Brandt, U (2006). “Energy converting NADH:quinone oxidoreductase (complex I)”. Annual Review of Biochemistry. 75: 69—92. DOI:10.1146/annurev.biochem.75.103004.142539. PMID 16756485.
- ↑ Grivennikova VG, Kotlyar AB, Karliner JS, Cecchini G, Vinogradov AD. (August 2007). “Redox-dependent change of nucleotide affinity to the active site of the mammalian complex I.”. Biochemistry. 46 (38): 10971—8. DOI:10.1021/bi7009822. PMC 2258335. PMID 17760425.
- ↑ Ермаков, 2005, с. 238.
- ↑ 1 2 Rozbeh Baradaran, John M. Berrisford, Gurdeep S. Minhas & Leonid A. Sazanov (28 February 2013). “Crystal structure of the entire respiratory complex I”. Nature. 494: 443–448. DOI:10.1038/nature11871.
- ↑ 1 2 3 Marion Babot, Amanda Birch, Paola Labarbuta, Alexander Galkin (July 2014). “Characterisation of the active/de-active transition of mitochondrial complex I”. Biochimica et Biophysica Acta (BBA) — Bioenergetics. 1837 (7): 1083—1092. DOI:10.1016/j.bbabio.2014.02.018.
- ↑ Galkin A, Moncada S (December 2007). “S-nitrosation of mitochondrial complex I depends on its structural conformation”. J. Biol. Chem. 282 (52): 37448—53. DOI:10.1074/jbc.M707543200. PMID 17956863.
- ↑ Moncada S, Erusalimsky JD (March 2002). “Does nitric oxide modulate mitochondrial energy generation and apoptosis?”. Nat. Rev. Mol. Cell Biol. 3 (3): 214—20. DOI:10.1038/nrm762. PMID 11994742.
- ↑ Lianwei Peng, Hiroshi Yamamoto, Toshiharu Shikanai (August 2011). “Structure and biogenesis of the chloroplast NAD(P)H dehydrogenase complex”. Biochimica et Biophysica Acta. 1807 (8): 945—953. DOI:10.1016/j.bbabio.2010.10.015.
- ↑ 1 2 Murphy MP (January 2009). “How mitochondria produce reactive oxygen species”. Biochem. J. 417 (1): 1—13. DOI:10.1042/BJ20081386. PMC 2605959. PMID 19061483.
- ↑ Hansford RG, Hogue BA, Mildaziene V (February 1997). “Dependence of H2O2 formation by rat heart mitochondria on substrate availability and donor age”. J. Bioenerg. Biomembr. 29 (1): 89—95. DOI:10.1023/A:1022420007908. PMID 9067806.
- ↑ Muller FL, Liu Y, Abdul-Ghani MA, Lustgarten MS, Bhattacharya A, Jang YC, Van Remmen H (January 2008). “High rates of superoxide production in skeletal-muscle mitochondria respiring on both complex I- and complex II-linked substrates”. Biochem. J. 409 (2): 491—9. DOI:10.1042/BJ20071162. PMID 17916065.
- ↑ Kussmaul L, Hirst J. (May 2006). “The mechanism of superoxide production by NADH:ubiquinone oxidoreductase (complex I) from bovine heart mitochondria”. Proceedings of the National Academy of Sciences of the United States of America. 103 (20): 7607—12. DOI:10.1073/pnas.0510977103. PMC 1472492. PMID 16682634.
- ↑ Esterházy D, King MS, Yakovlev G, Hirst J. (March 2008). “Production of reactive oxygen species by complex I (NADH:ubiquinone oxidoreductase) from Escherichia coli and comparison to the enzyme from mitochondria”. Biochemistry. 25 (12): 3964—71. DOI:10.1021/bi702243b. PMID 18307315.
- ↑ Moretti C, Grenand P (September 1988). “[The «nivrées», or ichthyotoxic plants of French Guyana]”. J Ethnopharmacol [фр.]. 6 (2): 139—60. DOI:10.1016/0378-8741(82)90002-2. PMID 7132401.
- ↑ 1 2 Watabe M, Nakaki T. (July 2008). “Mitochondrial complex I inhibitor rotenone inhibits and redistributes vesicular monoamine transporter 2 via nitration in human dopaminergic SH-SY5Y cells”. Molecular Pharmocology. 74 (4): 933—40. DOI:10.1124/mol.108.048546. PMID 18599602.
- ↑ Zharova TV, Vinogradov AD. (July 1997). “A competitive inhibition of the mitochondrial NADH-ubiquinone oxidoreductase (complex I) by ADP-ribose”. Biochimica et Biophysica Acta. 1320 (3): 256—64. DOI:10.1016/S0005-2728(97)00029-7. PMID 9230920.
- ↑ Nakamaru-Ogiso E, Han H, Matsuno-Yagi A, Keinan E, Sinha SC, Yagi T, Ohnishi T. (January 2010). “The ND2 subunit is labeled by a photoaffinity analogue of asimicin, a potent complex I inhibitor”. FEBS Letters. 584 (5): 883—8. DOI:10.1016/j.febslet.2010.01.004. PMC 2836797. PMID 20074573.
- ↑ Degli Esposti M, Ghelli A, Ratta M, Cortes D, Estornell E. (July 1994). “Natural substances (acetogenins) from the family Annonaceae are powerful inhibitors of mitochondrial NADH dehydrogenase (complex I)”. The Biochemical Journal. 301: 161—7. PMC 1137156. PMID 8037664.
- ↑ Viollet B, Guigas B, Sanz Garcia N, Leclerc J, Foretz M, Andreelli F. (March 2012). “Cellular and molecular mechanisms of metformin: an overview”. Clinical Science (London). 122 (6): 253—70. DOI:10.1042/CS20110386. PMID 22117616.
- ↑ Chou AP, Li S, Fitzmaurice AG, Bronstein JM. (April 2010). “Mechanisms of rotenone-induced proteasome inhibition”. Neurotoxicology. 113 (4): 674—82. DOI:10.1016/j.neuro.2010.04.006. PMC 2885979. PMID 20417232.
- ↑ Esteves AR, Lu J, Rodova M, Onyango I, Lezi E, Dubinsky R, Lyons KE, Pahwa R, Burns JM, Cardoso SM, Swerdlow RH. (February 2010). “Mitochondrial respiration and respiration-associated proteins in cell lines created through Parkinson’s subject mitochondrial transfer”. Journal of Neurochemistry. 113 (3): 674—82. DOI:10.1111/j.1471-4159.2010.06631.x. PMID 20132468.
- ↑ Andreazza AC, Shao L, Wang JF, Young LT. (April 2010). “Mitochondrial complex I activity and oxidative damage to mitochondrial proteins in the prefrontal cortex of patients with bipolar disorder”. Archives of General Psychiatry. 67 (4): 360—8. DOI:10.1001/archgenpsychiatry.2010.22. PMID 20368511.
- ↑ Moran M, Rivera H, Sánchez-Aragó M, Blázquez A, Merinero B, Ugalde C, Arenas J, Cuezva JM, Martín MA. (May 2010). “Mitochondrial bioenergetics and dynamics interplay in complex I-deficient fibroblasts”. Biochimica et Biophysica Acta. 1802 (5): 443—53. DOI:10.1016/j.bbadis.2010.02.001. PMID 20153825.
- ↑ Binukumar BK, Bal A, Kandimalla R, Sunkaria A, Gill KD. (April 2010). “Mitochondrial energy metabolism impairment and liver dysfunction following chronic exposure to dichlorvos”. Toxicology. 270 (2—3): 77—84. DOI:10.1016/j.tox.2010.01.017. PMID 20132858.
Литература
- Физиология растений / Под ред. И. П. Ермакова. — М.: Академия, 2005. — 634 с.
Внешние ссылки
- MRC MBU Sazanov group
- Complex I home page at The Scripps Research Institute
- MeSH Electron+Transport+Complex+I
| Статья является кандидатом в избранные статьи с 23 декабря 2015. |